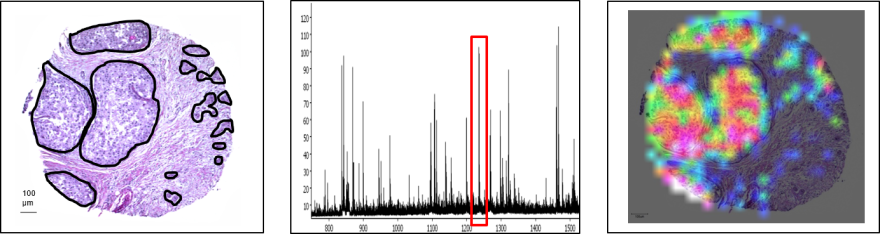
Bildgebende Massenspektrometrie
Die bildgebende Massenspektrometrie ist eine innovative Technologie um zahlreiche Peptide auf Gewebeschnitten zu identifizieren und deren Verteilung darzustellen.
In der Vergangenheit haben wir folgende Klassifikationsmodelle für die histopathologische Diagnostik entwickelt und publiziert:
- Amyloidose
- Hämophile Arthropathie
- Bewertung des Ausmaß der Degeneration des Meniskus
- Unterscheidung nicht-kleinzelliger Lungenkarzinome
- Unterscheidung zwischen chromophobem Nierenzellkarzinom und renalem Onkozytom
- Unterscheidung zwischen Melanom und Naevus
- Unterscheidung zwischen Adenokarzinom des Pankreas und neuroendokrinem Tumor des Pankreas
- Tumortypisierung von Mamma-, Pankreas-, Kolon- und Bronchialkarzinom
Referenzen:
[1] Anal Bioanal Chem. 2015 Jul; 407(18):5323-31. doi: 10.1007/s00216-015-8689-z.PMID: 25935672
[2] PLoS One. 2020 Jul 1; 15(7):e0235143. doi: 10.1371/journal.pone.0235143. PMID: 32609750
[3] Haemophilia. 2014 May; 20(3):446-53. doi: 10.1111/hae.12377. PMID: 24847521
[4] Z Rheumatol. 2015 Jun; 74(5):438-46. doi: 10.1007/s00393-014-1468-y. PMID: 25269875
[5] Mol Cell Proteomics. 2016 Oct; 15(10):3081-3089. doi: 10.1074/mcp.M115.057513. PMID: 27473201
[6] Cancers (Basel). 2020 Sep 21; 12(9):2704. doi: 10.3390/cancers12092704. PMID: 32967325
[7] J Cancer. 2020 Aug 21; 11(20):6081-6089. doi: 10.7150/jca.47698. PMID: 32922548
[8] Am J Dermatopathol. 2012 Feb; 34(1):82-90. doi: 10.1097/DAD.0b013e31823df1e2. PMID: 22197864
[9] Cancers (Basel). 2021 Jun 26;13(13):3197. doi: 10.3390/cancers13133197. PMID: 34206844
[10] Biochim Biophys Acta Proteins Proteom. 2017 Jul; 1865(7):858-864. doi: 10.1016/j.bbapap.2016.11.018. PMID: 27939606
[11] Proteomics. 2014 Apr; 14(7-8):956-64. doi: 10.1002/pmic.201300430. PMID: 24482424
[12] Proteomics Clin Appl. 2019 Jan; 13(1):e1800046. doi: 10.1002/prca.201800046. PMID: 30548962
[13] Oncotarget. 2020 Nov 3; 11(44):3998-4015. doi: 10.18632/oncotarget.27787. PMID: 33216824
[14] Proteomics Clin Appl. 2019 Jan; 13(1):e1700181. doi: 10.1002/prca.201700181. PMID: 30471200
[15] Int J Mol Sci. 2020 Aug 31; 21(17):6289. doi: 10.3390/ijms21176289. PMID: 32878024
[16] Proteomics Clin Appl. 2020 Nov; 14(6):e1900131. doi: 10.1002/prca.201900131. PMID: 32691971
Forschungsprojekte
Ci3
- Titel: Patientenzentrischer, integrierter Biomarker-, Forschungs- und Entwicklungshub
- Zusammenfassung: Ziel des Projektes war die patientenzentrierte Forschung mit integrierter Biomarkerforschungs unter Einbeziehung öffentlicher Forschungseinrichtungen, Transferinstituten und industriellen Partnern.
- Biomarkerforschungsdaten und Auswertung des klinischen Fortschritts bei 2500 Krebspatienten mit mindestens 5 verschiedenen Tumortypen (Lunge, Darm, Mamma, Ovar und Wiechteilsarkom). Diese Daten wurden in eine bioinformatische Plattform integriert als Kern für Nukleinsäureanalyse und immunologische Diagnostik
ZIM
- Titel: Entwicklung eines digitalen Färbeverfahrens als pathologisch-histologisches Diagnosewerkzeug auf Basis der MALDI Imaging Technologie
- Zusammenfassung: Ziel des Projektes war es einen digitalen Färbeprozess auf Basis der MALDI Imaging Technologie zu etablieren. Das digitale Bild soll dabei als alternative zu immunhistologischen Verfahren dienen. Weiterhin soll ein automatisierter Diagnosevorschlag für die histopathologische Diagnose erstellt werden. Es soll darüber hinaus geprüft werden, ob eine Malignitätsgraduierung möglich ist.
BMBF
- Titel: Massenspektrometrisches Profiling/Grading für onkologische Routineanwendungen der digitalen Pathologie (MaDiPath).
- Zusammenfassung: Ziel des Projektes war es, Methoden der digitalen Pathologie zu entwickeln, die eine objektive und reproduzierbare Basis für eine histopathologische Diagnose bilden und Therapieplanung mit dem Ziel immunhistologische Methoden zu unterstützen oder abzulösen. Dafür wurde ein top-down Lösungsansatz gewählt zur Klassifizierung von Tumordiagnosen basierend auf der Korrelation von tumorspezifischen Peptidmuster. Darüber hinaus wurden neue bioinformatische Methoden entwickelt, die eine Auswertung der generierten hochdimensionierten Daten erlauben.
BMBF
- Titel: Digitale Bildanalyse und bildgebende Massenspektrometrie zur Differenzierung von nicht-kleinzelligem Lungenkrebs - DIAMANT.
- Zusammenfassung: Dieses Projekt war auf die Kombination molekularer Informationen durch Massenspektrometrie mit der detailierten anatomischen Informationen des digitalen Gewebeschnittpräparats fokussiert, um multimodale deep-learning Algorithmen zur robusten Unterscheidung von Adenokarzinomen und Plattenepithelkarzinomen zu etablieren.